Deep Learning e Segmentação de Imagem na Próstata
Nesse post iremos ver como a segmentação de imagem baseada em deep learning pode ser usada para apoiar no diagnóstico de doenças prostáticas, proporcionando maior precisão, agilidade e suporte ao medi
Nos últimos anos, a segmentação de imagem com deep learning se destacou como uma técnica poderosa na medicina, especialmente no diagnóstico e acompanhamento de doenças na próstata. Este avanço é uma combinação de inteligência artificial e especialização médica, e tem potencial de transformar práticas clínicas. Neste post, vou explorar como a segmentação de imagem impacta a área da urologia e como os modelos de deep learning, como a U-Net, podem aprimorar diagnósticos e procedimentos terapêuticos.
O Papel da Segmentação de Imagem na Próstata
A próstata é uma glândula exócrina do sistema reprodutor masculino, localizada abaixo da bexiga e à frente do reto, circundando a uretra. Em média, possui o tamanho de uma noz e pesa entre 20 e 30 gramas.
Anatomicamente, a próstata é dividida em várias zonas:
Zona Central: Envolve os ductos ejaculadores.
Zona Periférica: A maior região da glândula e o local onde a maioria dos cânceres de próstata se desenvolve.
Zona de Transição: Circunda a uretra prostática e é comumente associada à hiperplasia prostática benigna (HPB).
Essas zonas, juntamente com o estroma fibromuscular composto por músculo e tecido fibroso, formam estruturas fundamentais para uma análise precisa de imagem.
A principal função da próstata é produzir um fluido que compõe cerca de 30% do sêmen, essencial para a sobrevivência dos espermatozoides. Este fluido é alcalino, ajudando a neutralizar a acidez da uretra e da vagina. Além disso, contém enzimas como o antígeno específico da próstata (PSA), zinco e ácido cítrico, que são cruciais para a motilidade e longevidade dos espermatozoides.
A próstata é regulada por hormônios, especialmente a testosterona, que é convertida em diidrotestosterona (DHT) dentro da glândula, desempenhando um papel importante em seu crescimento e funcionamento.
Dada a complexidade anatômica e funcional da próstata, sua análise exige alta precisão para diagnósticos e tratamentos eficazes de condições como câncer e hiperplasia benigna. A segmentação de imagem, utilizando tecnologia de deep learning, permite que médicos visualizem e avaliem, com precisão, áreas de interesse, como tecidos suspeitos. Essa tecnologia identifica automaticamente as bordas da próstata e outras estruturas relevantes, auxiliando na avaliação de tumores e na mensuração de volumes glandulares, o que aprimora a acurácia diagnóstica e o planejamento terapêutico.
Deep Learning em Ação: Modelos Convolucionais
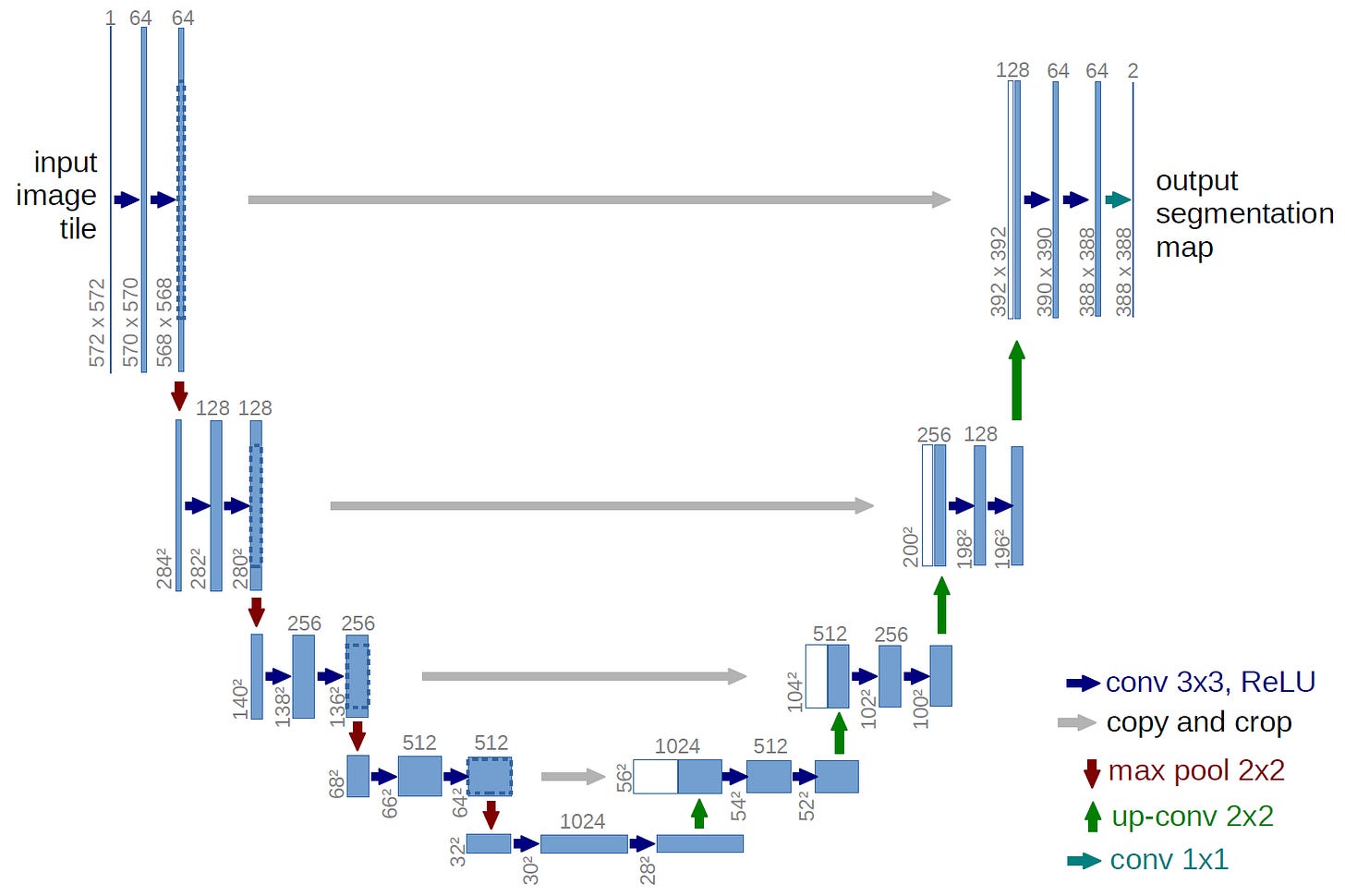
Modelos de deep learning, especialmente redes convolucionais como a U-Net, são amplamente utilizados para segmentar imagens médicas. A U-Net, projetada para segmentação semântica, possui uma arquitetura em formato de "U" composta por dois caminhos principais: o caminho de contração (encoder), que extrai características importantes reduzindo a resolução da imagem, e o caminho de expansão (decoder), que reconstrói a imagem com detalhes refinados. Essas etapas são interligadas por skip connections, que permitem preservar tanto o contexto global quanto os detalhes da imagem. Essa estrutura é particularmente eficiente para tarefas com dados rotulados limitados, comuns em imagens médicas, e oferece uma precisão que supera métodos tradicionais, sendo ideal para destacar áreas suspeitas na próstata.
Benefícios Clínicos da Segmentação Automatizada
Acurácia e Consistência: A análise computadorizada proporciona uma avaliação consistente e objetiva, minimizando a variabilidade humana, o que é fundamental no acompanhamento de câncer de próstata.
Rapidez no Diagnóstico: Modelos treinados podem fornecer resultados quase em tempo real, auxiliando em consultas ou mesmo em procedimentos minimamente invasivos guiados por imagem.
Suporte à Decisão Clínica: A segmentação precisa não só auxilia no diagnóstico inicial, mas também permite a monitoração da resposta ao tratamento, facilitando a adaptação terapêutica em tempo hábil.
Estrutura do Projeto para Segmentação de Imagens com U-Net
A seguir, apresento a estrutura de projeto utilizada para o desenvolvimento, treinamento e avaliação dos modelos de segmentação neste projeto. Essa organização facilita o gerenciamento do código e permite uma implementação eficiente e escalável da arquitetura U-Net.
project_root/
│
├── models/
│ ├── __init__.py
│ ├── unet.py # Define a arquitetura do modelo U-Net
│ ├── conv_block.py # Define o bloco convolucional reutilizável
│
├── data/
│ ├── __init__.py
│ ├── data_loader.py # Funções e classes para carregar e processar os dados # Dataset com as imagens de protosta e mascaras
│
├── utils/
│ ├── __init__.py
│ ├── metrics.py # Função para coeficiente de Dice e outras métricas
│ ├── plotting.py # Funções para visualização dos dados e métricas de treinamento
│
├── train.py # Script principal para treinar e avaliar o modelo
├── config.py # Configurações gerais do projeto (hiperparâmetros, paths)
├── requirements.txt # Dependências do projeto
└── README.md # Documentação do projeto
models/: Contém a arquitetura e componentes do modelo:
unet.py: Define a estrutura da U-Net.conv_block.py: Bloco convolucional reutilizável, componente essencial da U-Net.
data/: Lida com o carregamento e processamento de dados:
data_loader.py: Funções e classes para preparar os dados para o modelo.
utils/: Funções auxiliares:
metrics.py: Calcula métricas como o coeficiente de Dice, essencial para avaliar a qualidade da segmentação.plotting.py: Funções para visualização dos dados e acompanhamento das métricas durante o treinamento.
train.py: Script principal para treinar e avaliar o modelo, incluindo configuração de hiperparâmetros e execução do fluxo de treinamento.
config.py: Centraliza as configurações do projeto, como hiperparâmetros e caminhos para arquivos.
requirements.txt: Lista as dependências do projeto para fácil instalação.
README.md: Documentação do projeto com instruções de uso.
Mais abaixo desse post tem explicando de maneira resumida cada codigo
Dataset Utilizado no Projeto
Para este projeto de segmentação de imagem, utilizamos o dataset disponível em Zenodo que contém uma ampla variedade de imagens médicas com anotações específicas para treinamento e validação de modelos de segmentação. Este conjunto de dados é ideal para a tarefa, pois inclui imagens rotuladas que permitem treinar a U-Net para identificar com precisão as estruturas de interesse, como a próstata.
O dataset fornece uma base sólida para avaliação do modelo, permitindo que métricas como o coeficiente de Dice sejam calculadas para medir a acurácia da segmentação. A qualidade e diversidade das imagens tornam esse dataset uma escolha confiável para desenvolver modelos robustos e generalizáveis em segmentação de imagens médicas.
Estrutura da Parte de Modelos do Código
A pasta models contém os principais componentes para construção da arquitetura U-Net no projeto. Abaixo está uma explicação didática e resumida dos arquivos:
__init__.py:Este arquivo inicializa o módulo
modelse torna disponíveis as classesConv2dBlockeUNetWithResNetBackbonepara fácil importação em outras partes do projeto.
conv_block.py:Define a classe
Conv2dBlock, um bloco convolucional reutilizável, essencial para a construção da U-Net.Este bloco inclui duas camadas convolucionais, com opção de batch normalization para estabilizar o treinamento. Cada camada é ativada por uma função ReLU, facilitando a extração de características da imagem.
unet.py:Contém a classe
UNetWithResNetBackbone, que define a arquitetura principal do modelo U-Net com um backbone ResNet34.Encoder: Utiliza camadas da ResNet para extrair características importantes da imagem.
Decoder: Usa convoluções transpostas e o
Conv2dBlockpara reconstruir a imagem segmentada. Asskip connectionsconectam camadas do encoder com camadas do decoder para manter detalhes críticos.Funções de Salvar e Carregar: Inclui métodos para salvar e carregar o modelo, facilitando a continuidade do treinamento e uso do modelo em diferentes dispositivos.
Estrutura da Parte de Dados do Código
A pasta data contém o código responsável pelo carregamento e processamento dos dados de imagem, essencial para treinar e validar o modelo U-Net. Abaixo está uma explicação dos arquivos dessa pasta:
__init__.py:Inicializa o módulo
datae torna a funçãoload_datadisponível para importação em outras partes do projeto, facilitando o acesso ao carregamento de dados.
data_loader.py:Contém a função
load_data, responsável por carregar e preparar as imagens e máscaras para treinamento e validação do modelo.Processamento de Imagens: A função lê as imagens e máscaras, redimensiona-as para a dimensão especificada na configuração (
config.IM_HEIGHTeconfig.IM_WIDTH), converte para escala de cinza e normaliza os valores de pixel para o intervalo [0, 1].Divisão de Dados: Após o processamento, os dados são divididos em conjuntos de treinamento e validação usando
train_test_split, permitindo uma avaliação mais robusta do modelo.Transformação para Tensor: As imagens e máscaras são convertidas para tensores do PyTorch, organizadas no formato necessário (
(batch_size, channels, height, width)) e transferidas para o dispositivo especificado (config.DEVICE).
Estrutura da Parte de Utilitários do Código
A pasta utils contém funções auxiliares que ajudam na avaliação e visualização do desempenho do modelo, elementos essenciais para monitorar o treinamento e entender a qualidade da segmentação. Abaixo estão os detalhes dos arquivos dessa pasta:
__init__.py:Inicializa o módulo
utils, facilitando a importação das funçõesdice_coefeplot_learning_curveem outras partes do projeto.
metrics.py:Contém a função
dice_coef, que calcula o coeficiente de Dice, uma métrica essencial para avaliar a qualidade da segmentação.Cálculo do Dice Coefficient: A função achata as predições (
y_pred) e os valores reais (y_true), calcula a interseção entre eles e, em seguida, aplica a fórmula do coeficiente de Dice. Um valor próximo de 1 indica uma boa sobreposição entre a previsão e a verdade, essencial para avaliar modelos de segmentação.
plotting.py:Define a função
plot_learning_curve, que gera um gráfico de linha para visualizar a evolução das métricas de treinamento ao longo das épocas.Visualização das Métricas: A função exibe as curvas de perda de treinamento e validação, além do coeficiente de Dice ao longo do tempo. Isso ajuda a acompanhar o aprendizado do modelo e identificar possíveis problemas, como overfitting ou underfitting.
Estrutura do Código Principal do Projeto
Os arquivos config.py e train.py formam o núcleo do projeto, onde as configurações gerais são definidas e o fluxo de treinamento do modelo é implementado. Abaixo estão os detalhes de cada arquivo:
config.py:Contém as configurações gerais do projeto, facilitando ajustes rápidos de parâmetros:
DEVICE: Determina se o treinamento será executado em GPU (CUDA) ou CPU.
DATA_DIR: Caminho do diretório onde os dados de imagem estão armazenados.
IM_HEIGHT e IM_WIDTH: Altura e largura das imagens usadas no modelo.
N_FILTERS, DROPOUT: Parâmetros de configuração da U-Net.
BATCH_SIZE, LEARNING_RATE, NUM_EPOCHS: Parâmetros para o treinamento, como tamanho do lote, taxa de aprendizado e número de épocas.
MODEL_SAVE_PATH: Caminho para salvar o modelo treinado.
train.py:Define o processo de treinamento e validação do modelo, com as seguintes funções principais:
train: Controla o fluxo de treinamento por várias épocas, salvando as perdas de treinamento e validação, e calculando o coeficiente de Dice.
_train_step: Realiza um passo de treinamento individual, atualizando os pesos do modelo com base na perda.
validate: Avalia o modelo no conjunto de validação, calculando a perda média e o coeficiente de Dice para cada época.
predict: Faz previsões no conjunto de dados e aplica um limite para segmentar as predições.
plot_predictions: Visualiza imagens de exemplo, comparando a imagem original, a máscara real e a predição do modelo.
Execução Principal: O script inicializa o modelo U-Net com ResNet, o otimizador Adam e a função de perda BCE. Em seguida, carrega os dados de treinamento e validação, treina o modelo, salva os pesos treinados e realiza predições.
O repositorio desse projeto pode ser encontrado no meu github: https://github.com/FelipeAmaral13/ProjetosDeepLearning/tree/master/U_net_prostota_segmentation
Treinamento do modelo
Para treinar o modelo U-Net com backbone ResNet, utilizamos o dataset de imagens de ressonância magnética da próstata, dividido em conjuntos de treinamento e validação. O processo de treinamento foi configurado para otimizar o coeficiente de Dice, uma métrica importante para avaliar a qualidade da segmentação. Abaixo, detalhamos o processo de treinamento e os resultados obtidos.
Configuração do Treinamento
O treinamento foi realizado com as seguintes configurações:
Função de Perda: Utilizamos a função Binary Cross Entropy (BCE) para ajustar o modelo às máscaras binárias de segmentação.
Otimizador: O modelo foi otimizado com Adam, configurado com uma taxa de aprendizado adequada para garantir uma convergência estável.
Métricas: O coeficiente de Dice foi utilizado para avaliar a sobreposição entre as predições do modelo e as máscaras reais.
Curvas de Aprendizado
A primeira imagem mostra as curvas de perda de treinamento e validação ao longo das épocas, juntamente com a evolução do coeficiente de Dice. Observa-se uma queda inicial nas perdas, seguida por uma estabilização, o que indica que o modelo alcançou uma convergência razoável. No entanto, o coeficiente de Dice apresenta variações menores ao longo do treinamento, sugerindo que o modelo mantém uma performance consistente, mas com espaço para otimizações adicionais.
Resultados da Segmentação
Na segunda imagem, temos exemplos visuais dos resultados de segmentação. Para cada exemplo, exibimos:
Imagem Original: A imagem de entrada utilizada para a predição.
Máscara Real: A máscara de segmentação original, utilizada como referência.
Predição: A saída do modelo U-Net após o treinamento.
Esses exemplos demonstram que o modelo é capaz de identificar e segmentar a área da próstata com precisão razoável, embora algumas predições possam diferir ligeiramente das máscaras reais. Esse feedback visual é essencial para avaliar a qualidade do modelo e identificar possíveis melhorias.
O modelo U-Net, após o treinamento, mostrou-se eficaz na segmentação de imagens da próstata, obtendo boas métricas de sobreposição com as máscaras reais. As próximas etapas incluem ajustes finos nos hiperparâmetros e possivelmente o uso de técnicas de data augmentation para melhorar ainda mais a performance.
Conclusão e Trabalhos Futuros
A segmentação de imagem utilizando deep learning, especialmente com modelos como a U-Net, mostrou-se extremamente eficaz no diagnóstico e monitoramento de doenças na próstata. Com a arquitetura adequada e o uso de dados rotulados de qualidade, como o dataset utilizado neste projeto, foi possível alcançar resultados promissores, com boa precisão na identificação das áreas de interesse. Este avanço reforça o potencial da inteligência artificial para transformar práticas clínicas, oferecendo diagnósticos mais rápidos e consistentes, além de auxiliar no planejamento terapêutico.
Para aprimorar ainda mais os resultados, algumas melhorias podem ser implementadas:
Ajuste de Hiperparâmetros: Refinar parâmetros como a taxa de aprendizado, número de filtros e funções de ativação pode ajudar a melhorar a performance do modelo.
Data Augmentation: Introduzir técnicas de aumento de dados, como rotações e alterações de contraste, para expandir artificialmente o dataset, reduzindo o risco de overfitting e melhorando a generalização do modelo.
Exploração de Arquiteturas Avançadas: Testar variantes da U-Net ou arquiteturas mais complexas, como a Attention U-Net, que incorpora mecanismos de atenção para focar em áreas relevantes da imagem, pode aumentar a acurácia na segmentação.
Validação em Conjuntos de Dados Externos: Avaliar o modelo em outros datasets de imagens de próstata pode ajudar a verificar sua robustez e adaptabilidade em diferentes populações de pacientes.
Integração com Sistemas Clínicos: Desenvolver pipelines que integrem o modelo com sistemas de informação hospitalares para uso em tempo real na prática clínica.